BAO3 – Extraction de patrons morphosyntaxiques/relations de dépendances
La troisième Boite à Outils consiste à extraire des patrons morphosyntaxiques et des relations de dépendance à partir des fichiers que nous avons étiqueté lors de la Boite à Outils 2. Grâce au fichier BAO3.pl, nous allons pouvoir récupérer des patrons morphosyntaxique dans un fichier texte intitulé perlIsTheBigOne.txt. Ces patrons vont être comptés et classés par ordre descendant d'occurrences dans le fichier de sortie.
Code Perl : BAO3.pl – Extraction des patrons
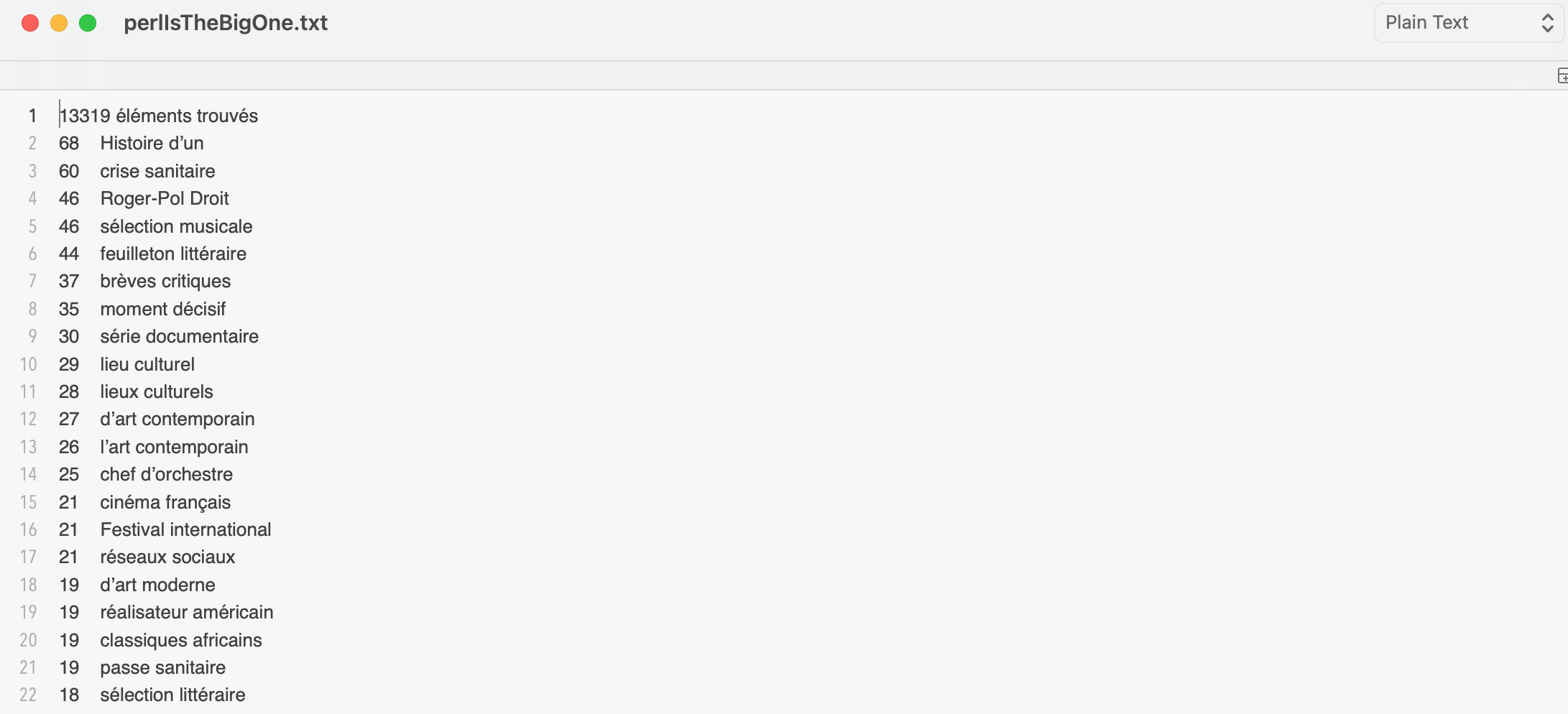
En entrée, ce script prend le fichier corpus-titre-description.xml ainsi que le patron morphosyntaxique recherché (ex. NOM ADJ, DET ADJ NOM, etc.) On lance le script de cette façon : BAO3.pl corpus-titre-description.xml [patron]. Ici, nous allons chercher les patrons NOM-ADJ : BAO3.pl corpus-titre-description.xml NOM ADJ. En sortie, nous récupérons le fichier perlIsTheBigOne.txt.
Et voila à quoi ressemble le fichier de sortie :
Code Perl : extract-relation-udpipe.pl – Extraction des relations de dépendances
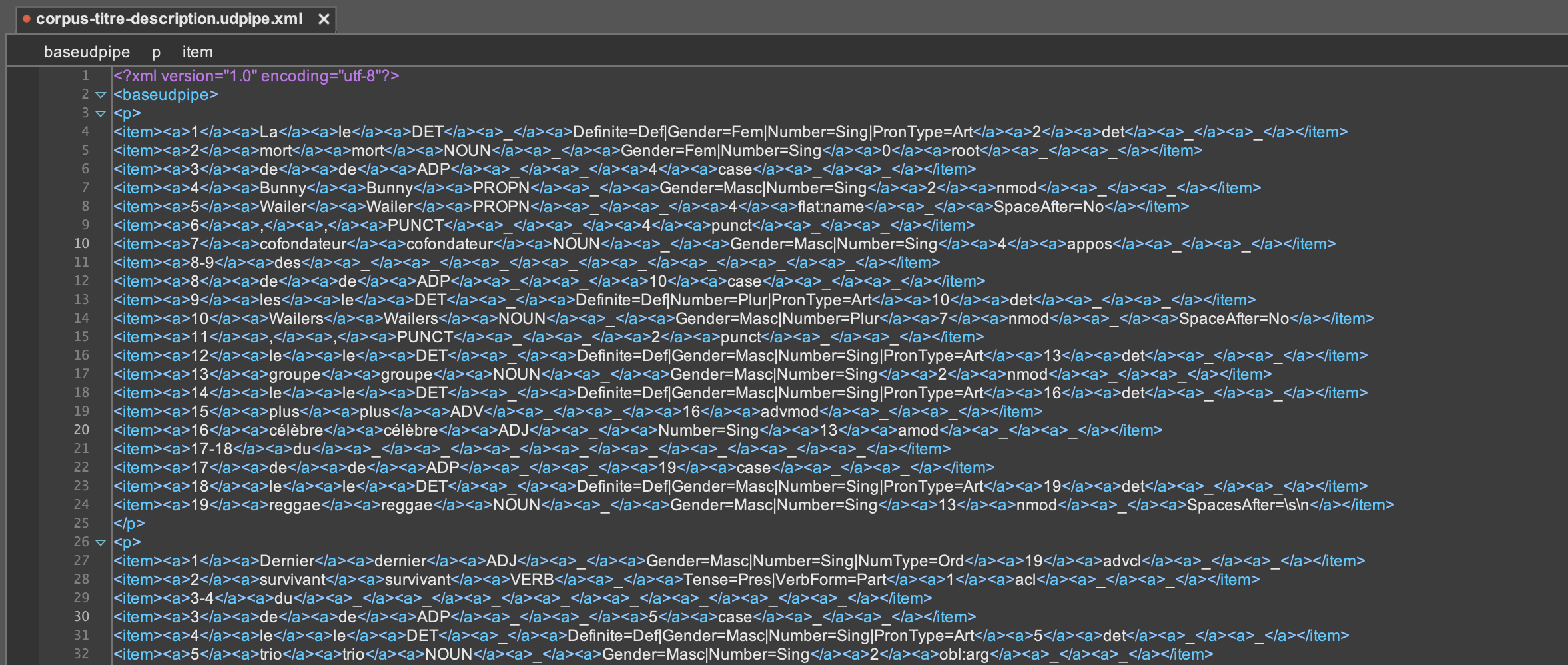
On peut ensuite utiliser le fichier corpus-titre-description.udpipe que nous avons créer lors de la BàO2 et le mettre au format XML grace au fichier udpipe2xml.pl : perl udpipe2xml.pl corpus-titre-description.udpipe. En sortie, nous obtenons un fichier XML :
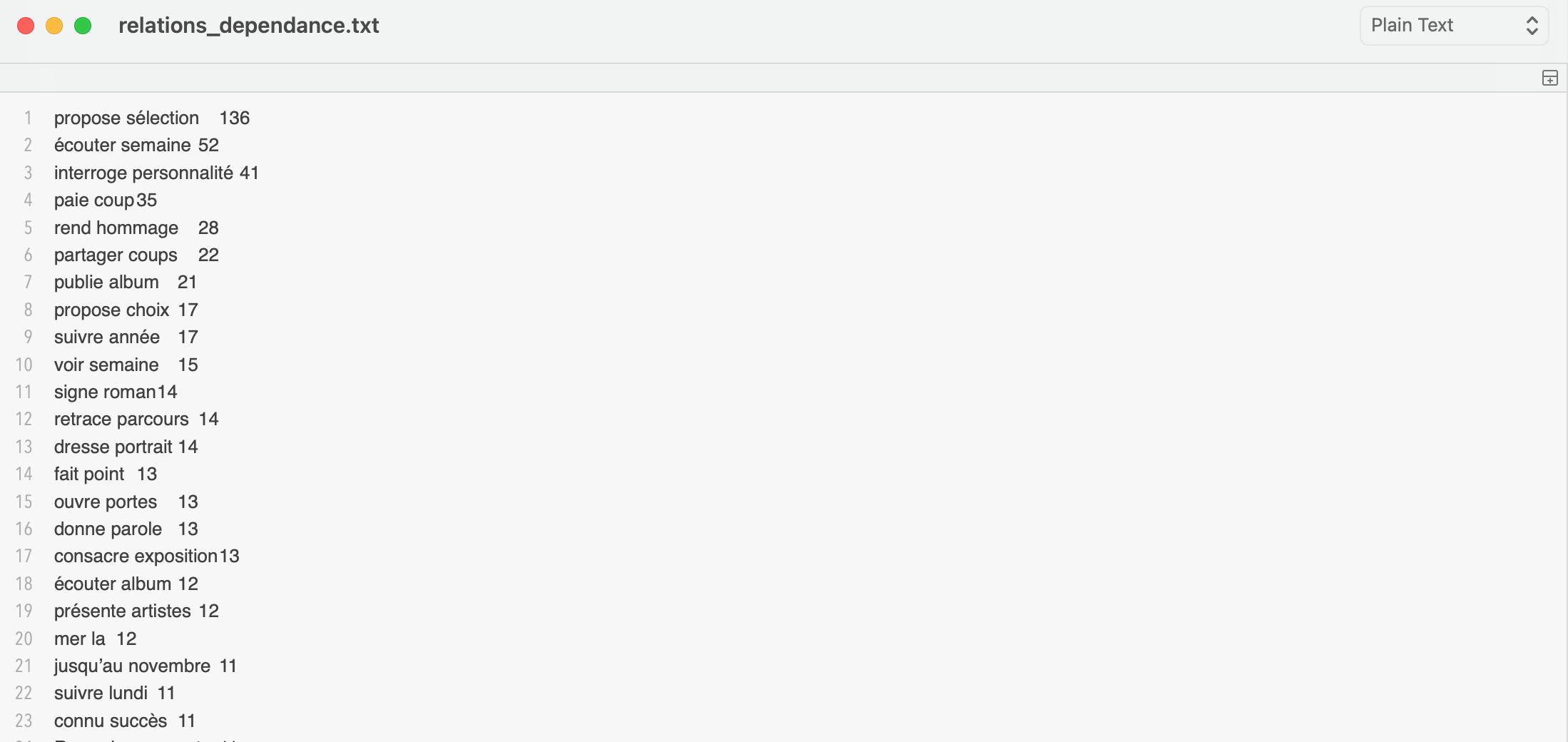
Nous pouvons ensuite utiliser notre nouveau fichier corpus-titre-description.udpipe.xml afin de récupérer les relations de dépandances. Pour cela, nous utilisons le script extract-relation-udpipe.pl. Dans le terminal, nous pouvons écrire : perl extract-relation-udpipe.pl corpus-titre-description.udpipe.xml [relation] > [sortie]. Ici, nous avons choisit la relation objet ("obj") et notre fichier de sortie sera relations_dependance.txt : perl extract-relation-udpipe.pl corpus-titre-description.udpipe.xml "obj" > relations_dependance.txt.
Et voici le fichier relations_dependance.txt :
Code Python
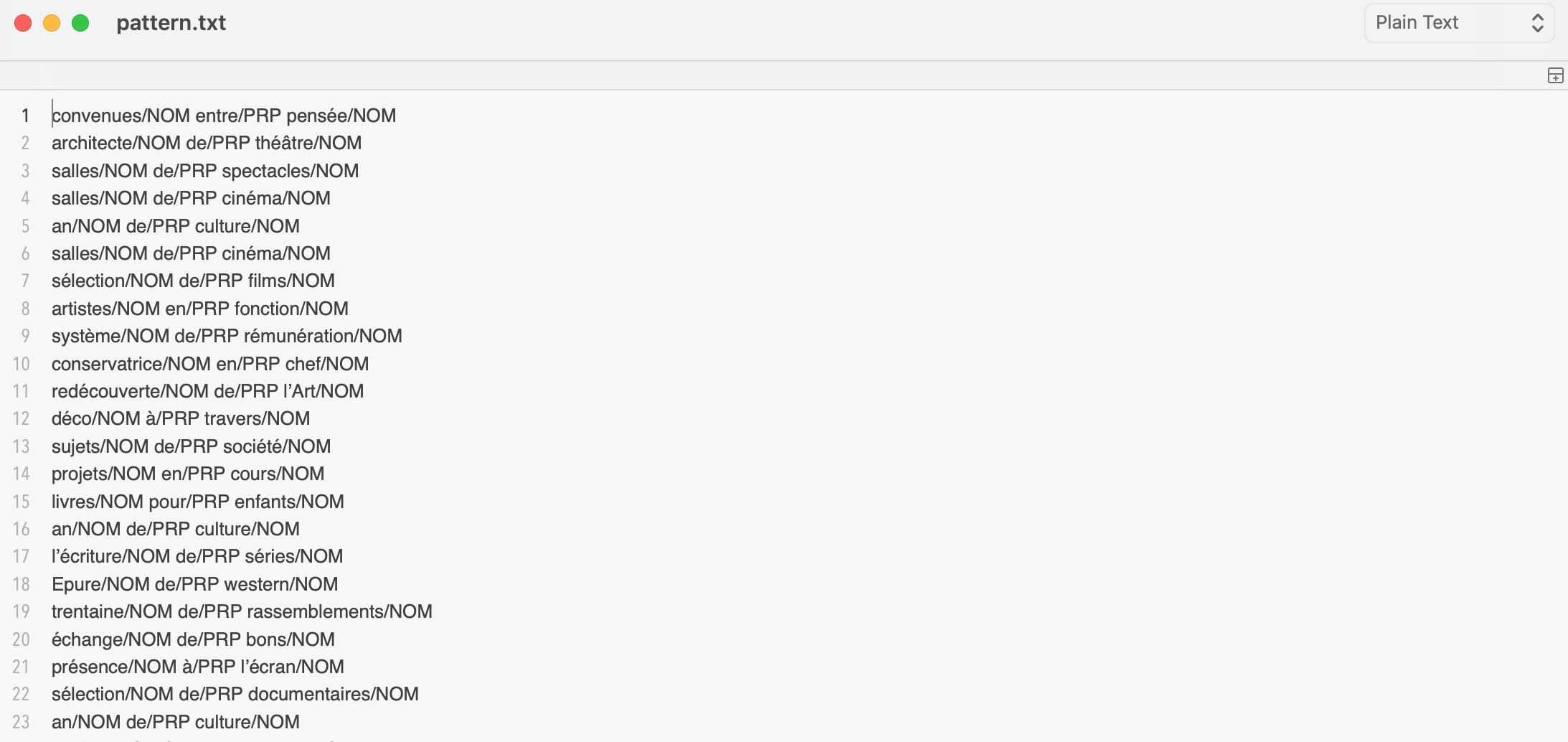
Pour cette BàO3, le script Python nous permet d'effectuer la même tâche que le script Perl BAO3.pl : il nous permet de récupérer des patrons morphosyntaxiques à partir de notre fichier corpus-titre-description.xml. Ce script se lance de cette façon : python3 BAO3.py corpus-titre-description.xml [patron] > [sortie]. Ici, nous allons chercher les patrons NOM-PRP-NOM et mettre un fichier de sortie nommé pattern.txt : python3 BAO3.py corpus-titre-description.xml NOM PRP NOM > pattern.txt.
Voici un apperçu du fichier texte :