Programmation et Projet encadré

Je m'appelle LIU Pan, étudiante en Master 1 Traitement Automatique des Langues, cohabilité par l'Institut National des Langues et Civilisations Orientales (INALCO), Paris 3 (Sorbonne Nouvelle) et Paris-Nanterre.
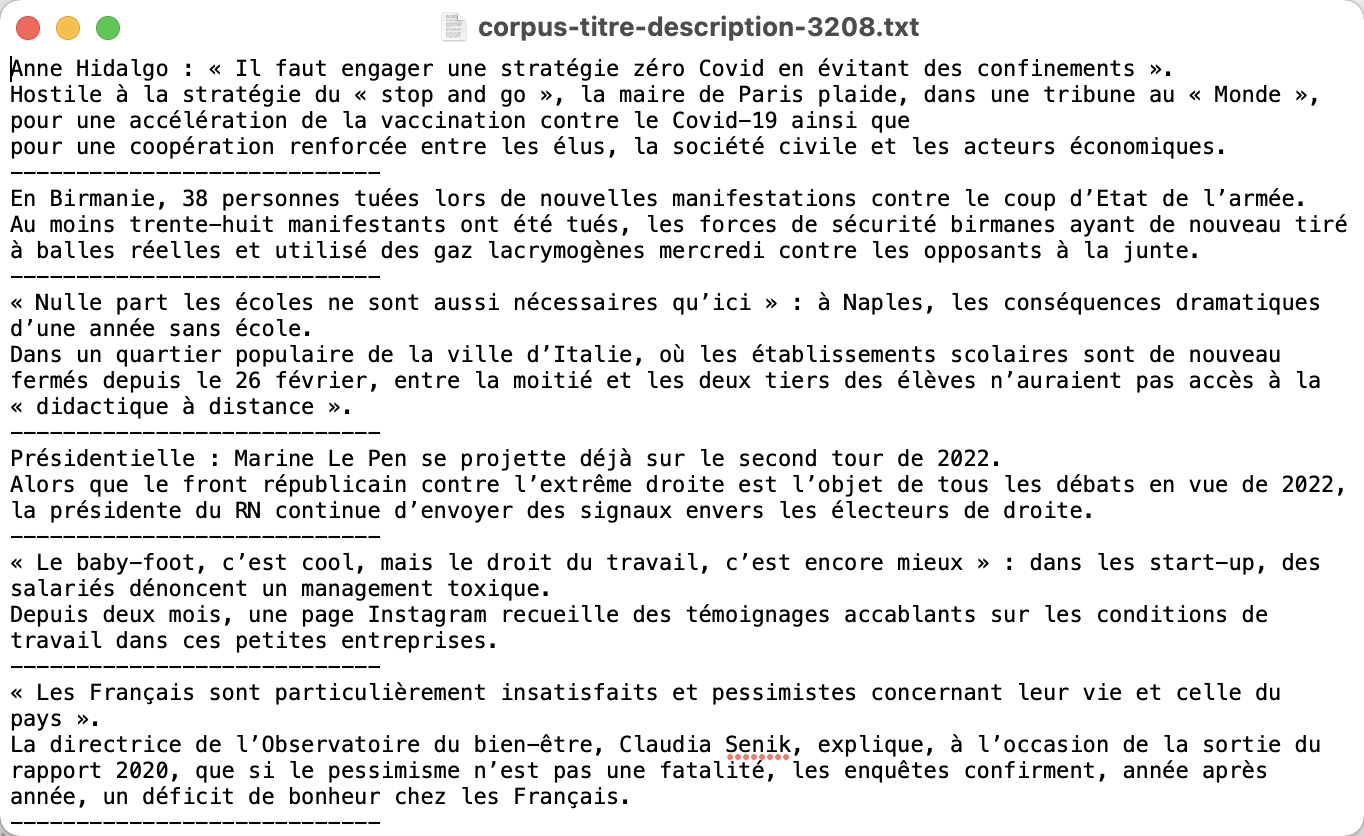
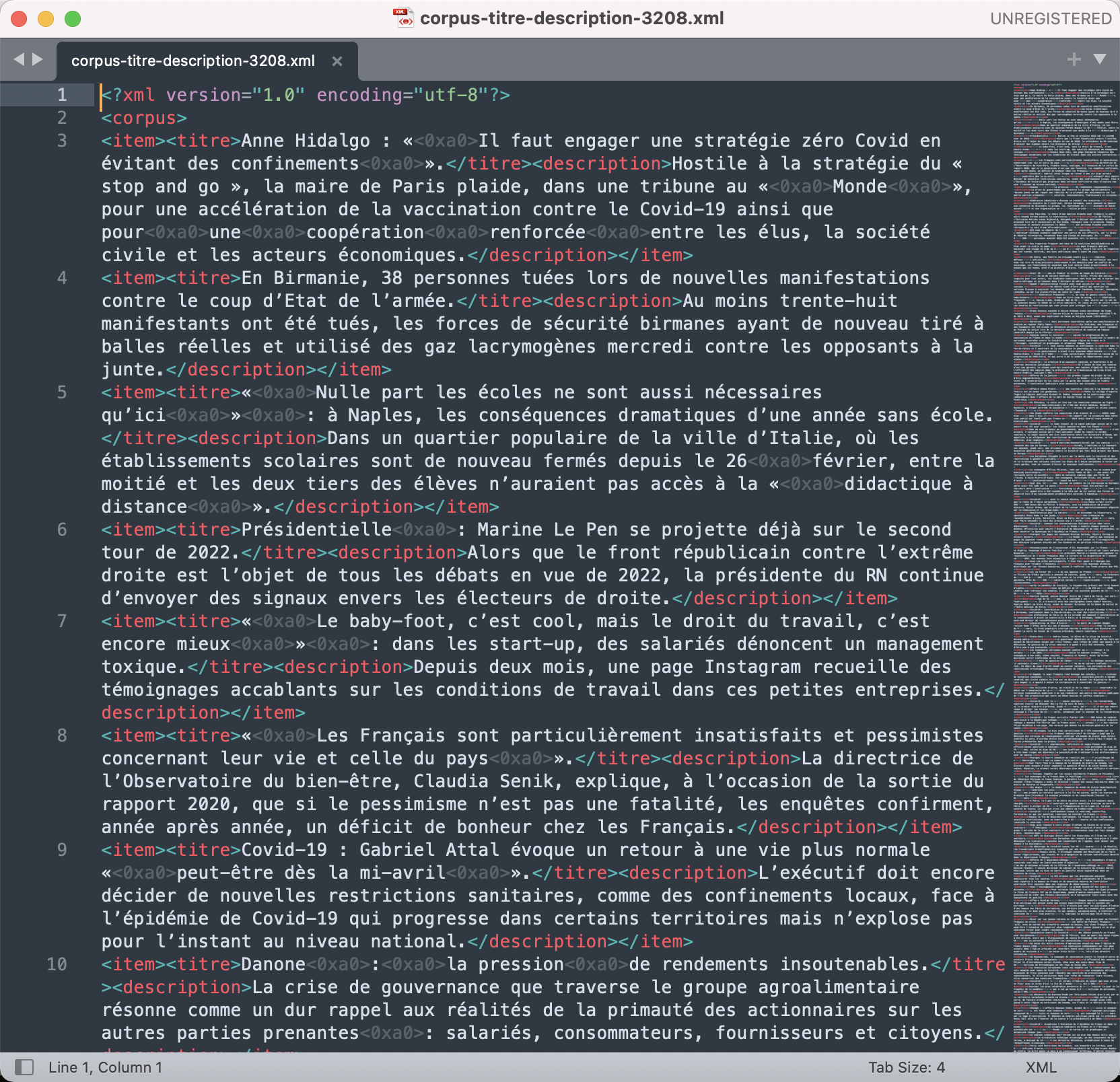
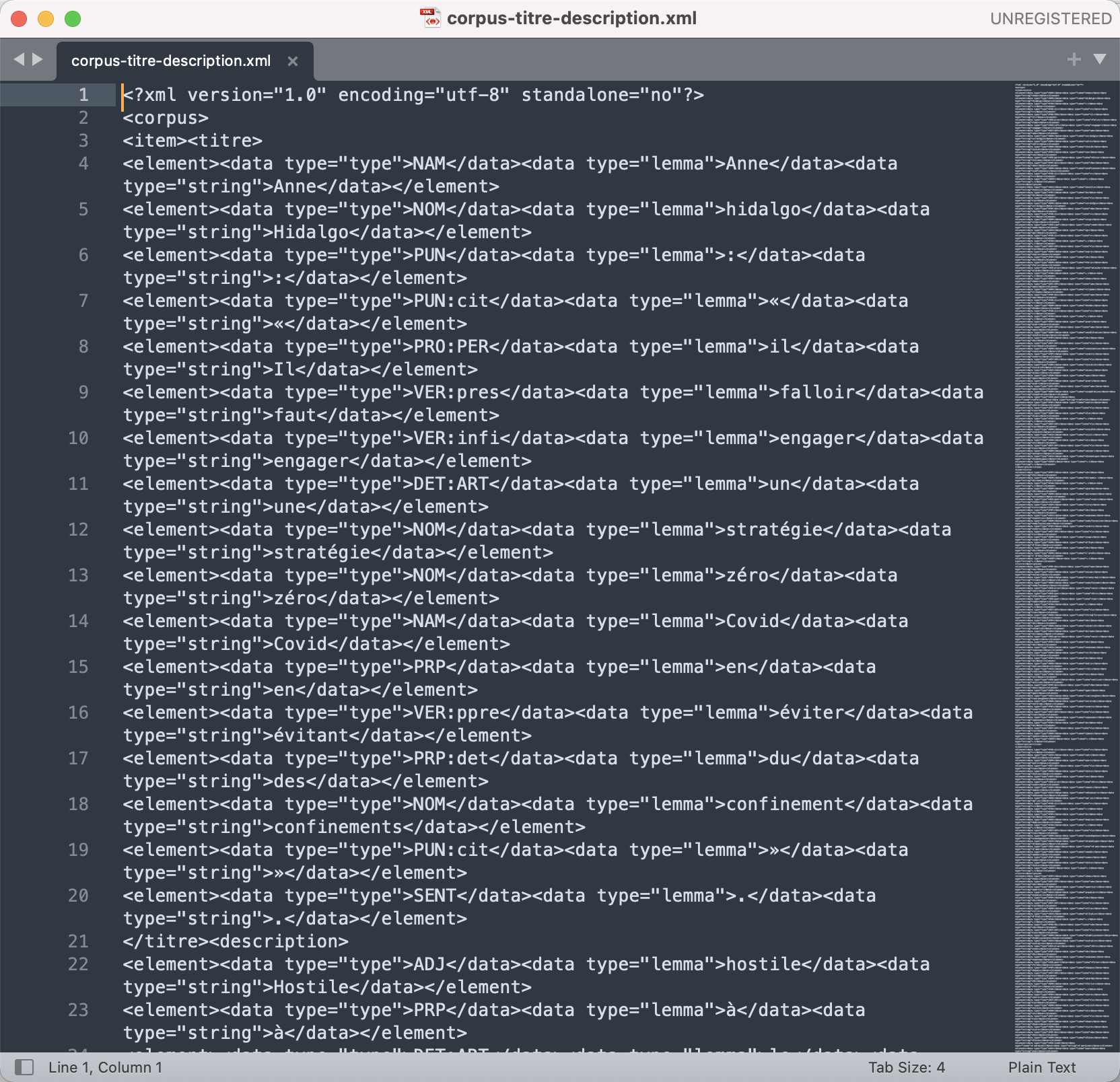
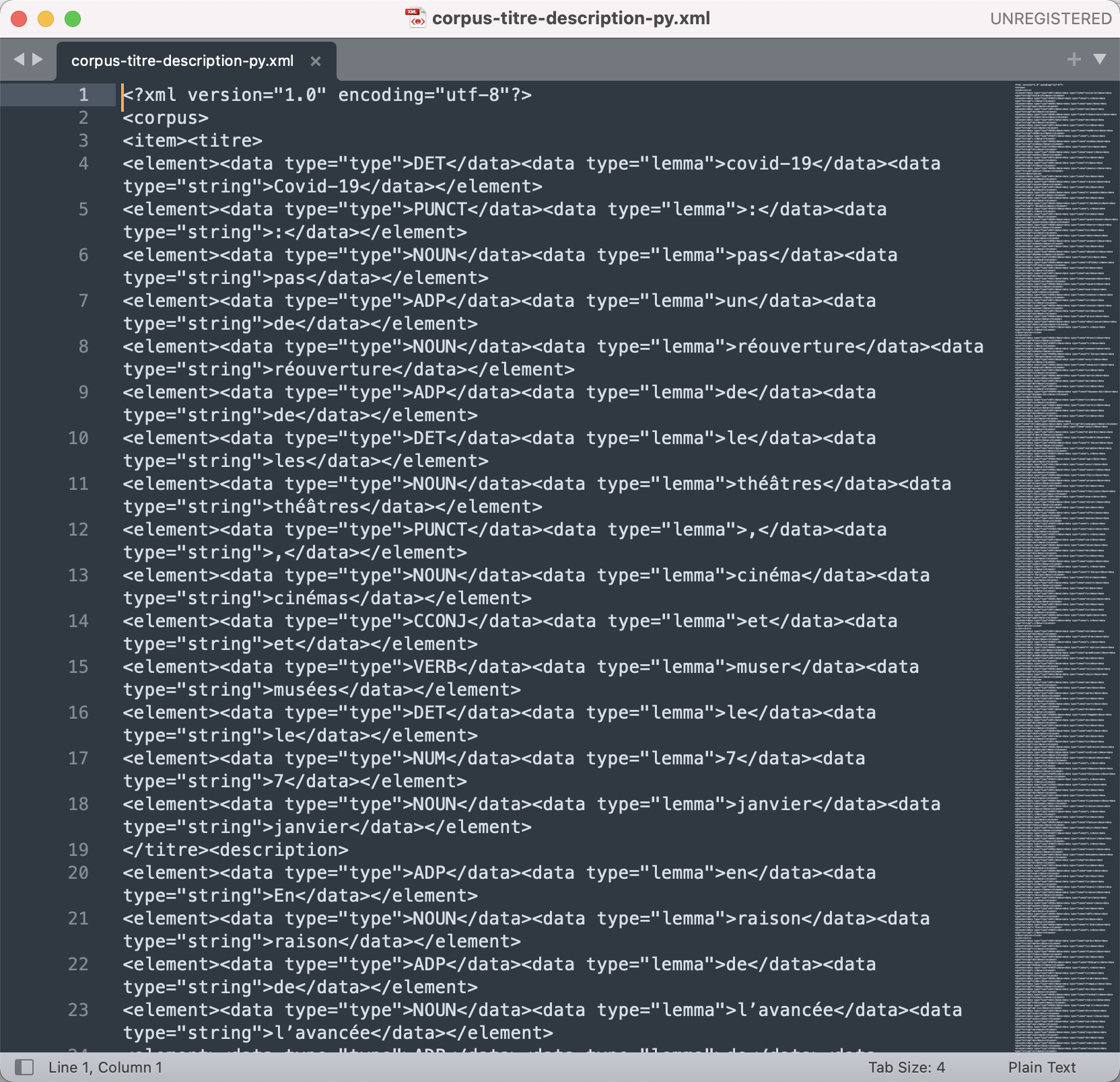
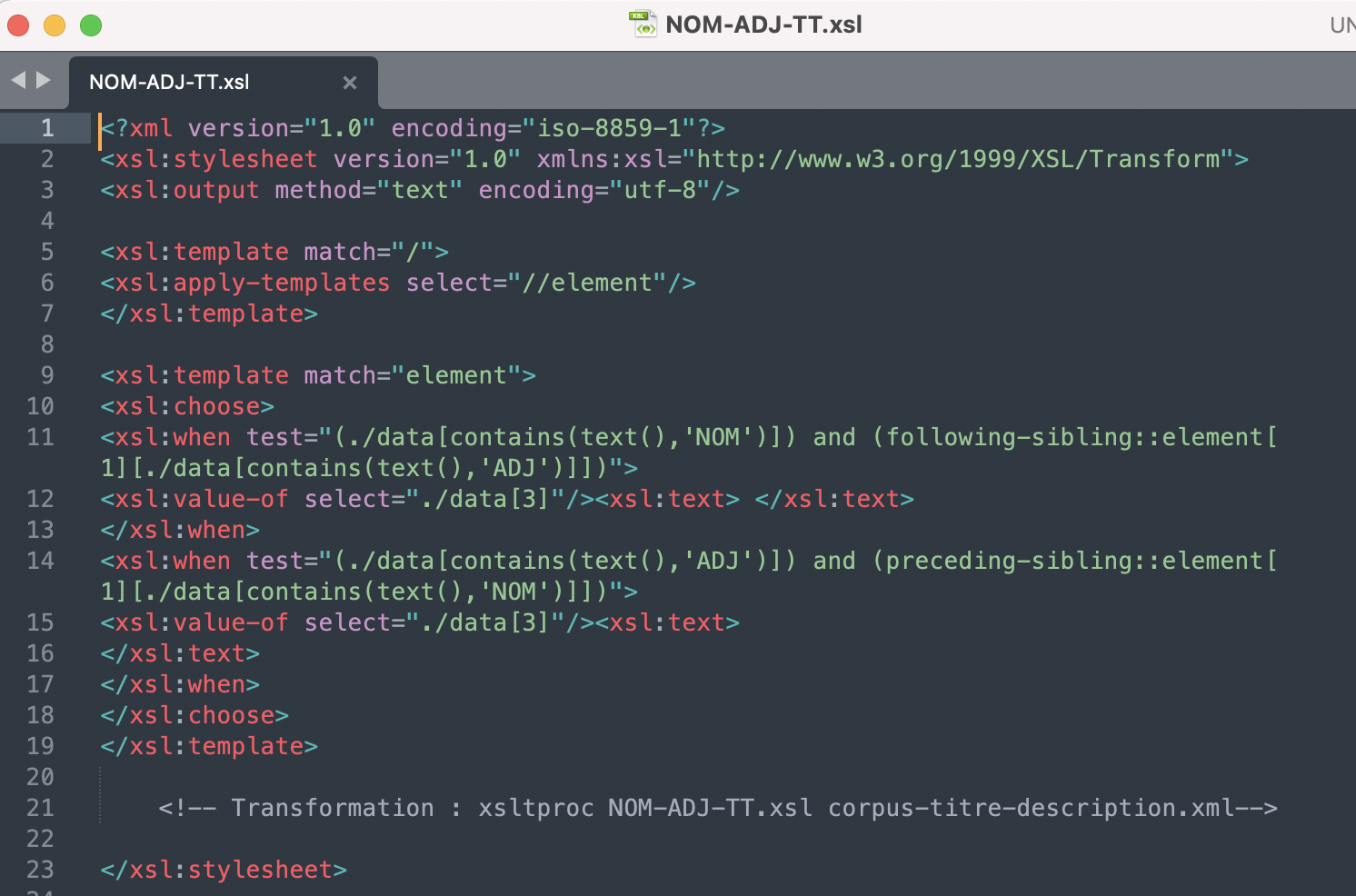
Le projet des boites à outils a pour but d'extraire les titres et description d'un flux RSS afin de les étiqueter et de faire de l'extraction terminologique à partir de patron. Il a également pour but l’apprentissage du langage Perl.
Le corpus de travail sera constitué de l'ensemble des fils RSS disponibles sur le site du journal Le Monde recueillis tous les jours de l'année 2021 à 19h.