Prorgrammation
On garde la même structure de programme que celle de BAO1 en ajoutant, après avoir parcouru l'arborescence, les fonctions qui appellent Treetagger et UDpipe, c'est à dire d'utiliser la fonction de perl system() pour lancer les commandes bash.
#obtenir l'extraction pour trois rubriques, en parcourant la liste des rubriques
foreach my $r (@RUBRIQUES){
# créer un dictionnaire vide qui enregistre les informations distingues (sans répétition) pour chaque rubrique
my %dico_des_titres=();
# créer un nouveau fichier .txt et un nouveau fichier .xml qui contient le numéro de rubrique
open my $out, ">:encoding(UTF-8)","sortie-slurp_$r.txt";
open my $outxml, ">:encoding(UTF-8)","sortiexml-slurp_$r.xml";
print $outxml "<?xml version=\"1.0\" encoding=\"utf-8\" ?>>\n";
print $outxml "<corpus2020>\n";
# parcourir l'arborescence et trouver les informations dans la rubrique ciblée
# en portant les paramètres nom de l'arborescence, le numéro de rubrique, le fichier .txt, le fichier .xml,
# et le dictionnaire qui mémorise les informations déjà enregistrées
&parcoursarborescencefichiers($rep,$r,$out,$outxml,%dico_des_titres);
print $outxml "</corpus2020>\n";
close $out;
close $outxml;
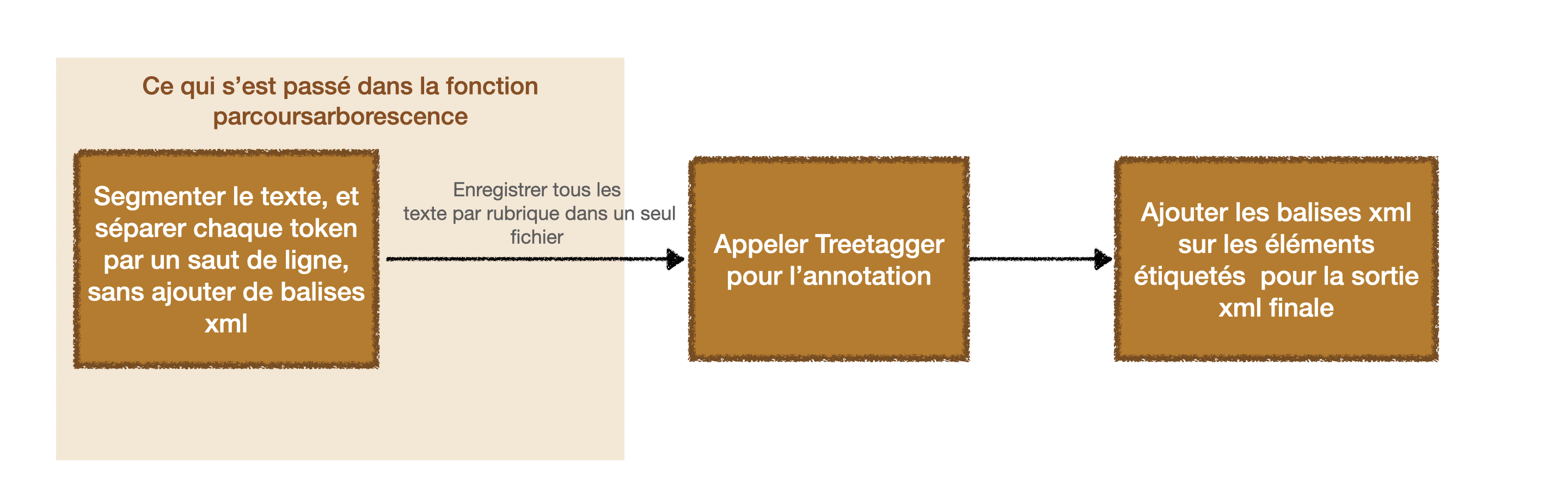
# appler la fonction d'étiquetagge TreeTagger
# en donnant un paramètre pour il trouve le bon fichier grâce au repère de rubrique
&etiquetageTT($r);
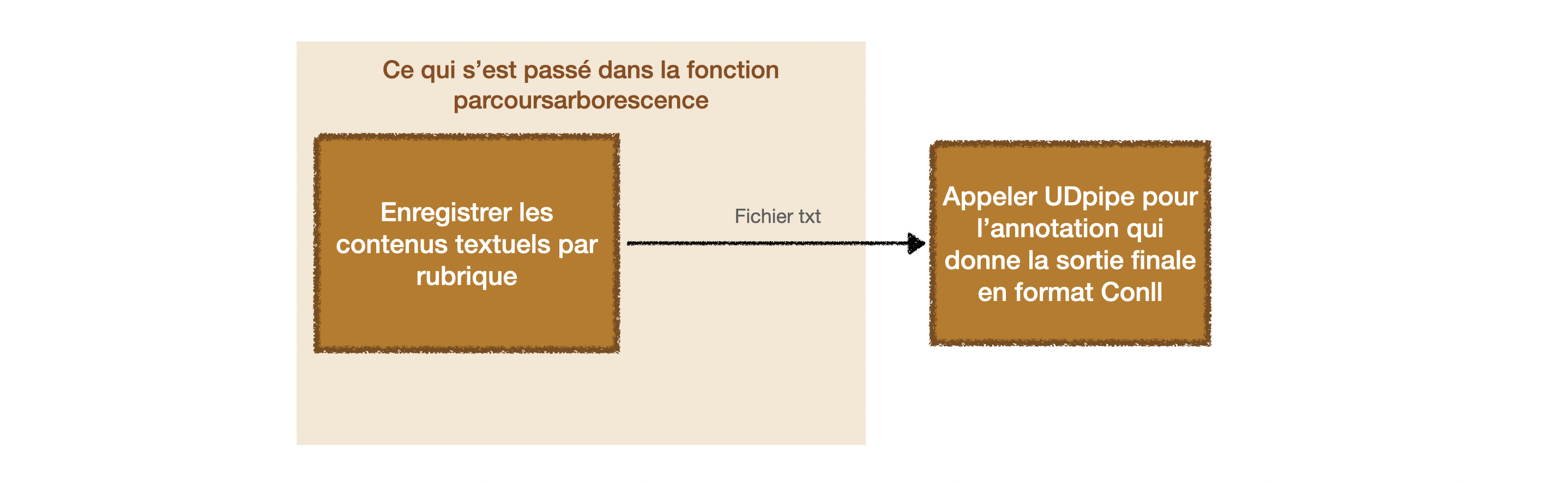
# appler la fonction d'étiquetagge Udpipe
# en donnant un paramètre pour il trouve le bon fichier grâce au repère de rubrique
&etiquetageUD($r);
}
#-----------------------------------------
sub etiquetageTT {
my $rubrique = shift(@_);
# lancer tree-tagger-MacOSX-3.2.3
system ("./tree-tagger-MacOSX-3.2.3/bin/tree-tagger ./tree-tagger-MacOSX-3.2.3/bin/french-utf8.par -token -lemma -no-unknown -sgml sortiexml-slurp_$rubrique.xml > sortiexml-slurp_TT_$rubrique.txt");
system ("perl ./tree-tagger-MacOSX-3.2.3/bin/treetagger2xml-utf8.pl sortiexml-slurp_TT_$rubrique.txt utf8");
}
sub etiquetageUD {
my $rubrique = shift(@_);
# Etiquetage avec udpipe
# lancer udpipe : distrib-udpipe-1.2.0-bin
system("./distrib-udpipe-1.2.0-bin/udpipe-1.2.0-bin/bin-osx/udpipe --tokenize --tag --parse ./distrib-udpipe-1.2.0-bin/modeles/french-gsd-ud-2.5-191206.udpipe sortie-slurp_$rubrique.txt > sortieudpipe-slurp_$rubrique.txt");
}
Pour faire la tokenization(tous les tokens séparés par un saut de ligne) pendant le parcours de l'arborescence, on doit appeler une fonction qui s'appelle preetiquetage()
sub preetiquetage {
my $titre = $_[0];
my $description = $_[1];
#-----------------etiquetage titre-----------------------------
# créer un fichier temporaire.txt et test.txt.pos pour enregistrer temporarement les informations
# parce que Treetagger ne peut pas traiter directement les chaines de caractères, il faut prendre les inforamtions à partir d'un fichier
open (ETI, ">:encoding(utf8)", "temporaire.txt");
print ETI $titre;
close ETI;
system ("perl -f ./tree-tagger-MacOSX-3.2.3/bin/tokenise-utf8.pl temporaire.txt > test.txt.pos");
open (TEMP, "<:encoding(utf8)", "test.txt.pos");
$/=undef;
my $titre_etik_xml=<TEMP>;
close TEMP;
#-----------------etiquetage description-----------------------------
open (ETI, ">:encoding(utf8)", "temporaire.txt");
print ETI $description;
close ETI;
system ("perl -f ./tree-tagger-MacOSX-3.2.3/bin/tokenise-utf8.pl temporaire.txt > test.txt.pos");
open (TEMP, "<:encoding(utf8)", "test.txt.pos");
$/=undef;
my $description_etik_xml=;
close TEMP;
return $titre_etik_xml,$description_etik_xml;
}